 いろいろな病気は、たん白質の異常作用が原因となって生じると考えられており、最近の新薬開発ではこうした異常なたん白質に結合してその働きをコントロールする分子を発見することがターゲットになってきている。つまり、そうした標的たん白質に最もよく結合する分子が医薬品の重要な候補となるわけで、最近ではこのためのドッキングシミュレーションが広く関心を集めている。
いろいろな病気は、たん白質の異常作用が原因となって生じると考えられており、最近の新薬開発ではこうした異常なたん白質に結合してその働きをコントロールする分子を発見することがターゲットになってきている。つまり、そうした標的たん白質に最もよく結合する分子が医薬品の重要な候補となるわけで、最近ではこのためのドッキングシミュレーションが広く関心を集めている。
富士通と東海大学が創薬支援計算化学で実証実験に成功
分子軌道法によるドッキングシミュレーションを実施、実験の置き換えも可能に
2003.11.11−富士通と東海大学は11日、コンピューターを利用して新薬の探索を行う計算化学技術の実証実験に成功したと発表した。半経験的分子軌道法の高速計算手法である“”ローカルSCF法”(局所自己無撞着場法)を世界で初めてプログラム化し、HIVプロテアーゼと実際に臨床試験中の数種類の阻害剤とのドッキングシミュレーションを実施したもの。精度と計算時間が実用域に達したことから、動物実験などの削減を含め、新薬開発の時間と経費を節減できることが実際に確認できたとしている。富士通では、今回のプログラムを「CAChe localSCF」として製品化し、全世界の製薬会社などに向けて売り込んでいく。
 いろいろな病気は、たん白質の異常作用が原因となって生じると考えられており、最近の新薬開発ではこうした異常なたん白質に結合してその働きをコントロールする分子を発見することがターゲットになってきている。つまり、そうした標的たん白質に最もよく結合する分子が医薬品の重要な候補となるわけで、最近ではこのためのドッキングシミュレーションが広く関心を集めている。
いろいろな病気は、たん白質の異常作用が原因となって生じると考えられており、最近の新薬開発ではこうした異常なたん白質に結合してその働きをコントロールする分子を発見することがターゲットになってきている。つまり、そうした標的たん白質に最もよく結合する分子が医薬品の重要な候補となるわけで、最近ではこのためのドッキングシミュレーションが広く関心を集めている。
しかし、「現在は古典力学をベースとしたシミュレーションがほとんどで、精度に問題があり、電子の影響を考慮できないために活性部位で共有結合を形成するような系はまったく扱うことができなかった。一方で、量子力学をベースにした分子軌道法は時間がかかりすぎて実用的ではなかった」(東海大学医学部の平山令明教授)という。
今回の富士通と東海大学の共同研究は、標的たん白質と医薬候補化合物とのドッキングシミュレーションを量子力学的に行えるようにしたことがポイント。“ローカルSCF法”は半経験的分子軌道法における最新の高速計算手法で、プログラム化されたのは世界でも初めてだという。富士通には、MOPAC2002に採用されている“MOZYME法”もあるが、今回の“ローカルSCF法”は一段の高速化を達成。ベンチマークでは、3,686原子からなるバクテリオロドプシンを対象にしたCPUタイムは、MOZYME法で3,064秒、ローカルSCF法は1,083秒となった。使用メモリー量もMOZYME法の512メガバイトに対してローカルSCF法は55メガバイトと小さい。11万原子を持つシャペロニンの場合は、同様に146時間と12時間、88ギガバイトと1.2ギガバイトという結果だった。
今回の実証実験においては、抗エイズウイルス薬の標的であるHIV-1プロテアーゼに対して、現在臨床的に使用されている4種類の阻害剤がどのように相互作用するかをシミュレーションで検証した。その一例においては、3,095原子の複合体に対し、ペンティアム4(2.8GHz)/2ギガバイトメモリーのパソコンを用いて3時間44分でその相互作用エネルギーを計算することができた。同じものを同じマシンでMOZYME法で計算したケースでは約120時間が必要だった。計算結果と実験データとの間にも良い相関がみられたという(実験によって得られた解離速度常数と計算で求めた相互作用エネルギーとが定性的に一致した/写真参照)。
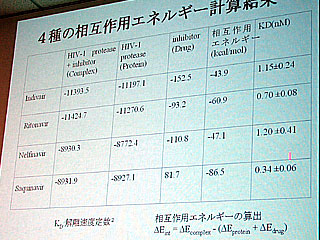 平山教授は、「たん白質の構造や生物活性を扱う意味では十分な精度に達していると思う。計算時間との比較でも、精密なアッセイを行う場合とほぼ同等のスループットをトータルで出せそうだ」と話す。共同研究グループでは、実験をある程度置き換えることが現実に可能であり、今後の創薬研究の新しい可能性を開くことができたとしている。今後はとくに、緑内障治療薬の一種のように活性部位で共有結合を起こすような医薬を対象に計算に取り組みたいという。
平山教授は、「たん白質の構造や生物活性を扱う意味では十分な精度に達していると思う。計算時間との比較でも、精密なアッセイを行う場合とほぼ同等のスループットをトータルで出せそうだ」と話す。共同研究グループでは、実験をある程度置き換えることが現実に可能であり、今後の創薬研究の新しい可能性を開くことができたとしている。今後はとくに、緑内障治療薬の一種のように活性部位で共有結合を起こすような医薬を対象に計算に取り組みたいという。
なお、今回のプログラムは「CAChe localSCF」として製品化される。CACheの名を冠しているが、当面は純粋なソルバーコードとして提供し、CACheのグラフィック環境と統合するのは次期バージョンからになる模様。