 VoyaGeneは、MKIが新エネルギー・産業技術総合開発機構(NEDO)のゲノムインフォマティクスプロジェクトの一環として委託を受けて1998年度から実施してきたプロジェクトで、東京理化大学の村上康文教授らのグループとの共同研究を背景として開発を進めてきた。また、九州大学の久原哲教授および岡本正宏教授からデータ面での協力を得た。プロジェクトはこの3月末で終了し、4月から事業化のフェーズに入る。
VoyaGeneは、MKIが新エネルギー・産業技術総合開発機構(NEDO)のゲノムインフォマティクスプロジェクトの一環として委託を受けて1998年度から実施してきたプロジェクトで、東京理化大学の村上康文教授らのグループとの共同研究を背景として開発を進めてきた。また、九州大学の久原哲教授および岡本正宏教授からデータ面での協力を得た。プロジェクトはこの3月末で終了し、4月から事業化のフェーズに入る。
三井情報開発が遺伝子ネットワーク同定研究支援システムを開発
4種類のネットワークモデルを駆使、2003年度内に製品化へ
2003.03.25−三井情報開発(MKI)は、遺伝子発現のメカニズム解明に役立つ遺伝子ネットワーク同定研究支援システム「VoyaGene」(ヴォイジーン)を開発した。2003年度内に製品化して売り出す計画で、昨年10月からインターネットでベータ版を公開し、ユーザーの意見や要望を集めている。とくに、遺伝子ネットワークの複数の予測手法を組み合わせて利用できるシステムは世界でも初めてだとしており、ポストゲノム研究の強力な武器になると期待されている。
 VoyaGeneは、MKIが新エネルギー・産業技術総合開発機構(NEDO)のゲノムインフォマティクスプロジェクトの一環として委託を受けて1998年度から実施してきたプロジェクトで、東京理化大学の村上康文教授らのグループとの共同研究を背景として開発を進めてきた。また、九州大学の久原哲教授および岡本正宏教授からデータ面での協力を得た。プロジェクトはこの3月末で終了し、4月から事業化のフェーズに入る。
VoyaGeneは、MKIが新エネルギー・産業技術総合開発機構(NEDO)のゲノムインフォマティクスプロジェクトの一環として委託を受けて1998年度から実施してきたプロジェクトで、東京理化大学の村上康文教授らのグループとの共同研究を背景として開発を進めてきた。また、九州大学の久原哲教授および岡本正宏教授からデータ面での協力を得た。プロジェクトはこの3月末で終了し、4月から事業化のフェーズに入る。
パッケージソフトのかたちで2003年度内に製品化する予定だが、個別のカスタマイズをベースにしたソリューションビジネスとしての受注・構築活動には直ちに着手する考えであり、公開中のベータ版はそのためのアンテナとしての役割も果たしているという。
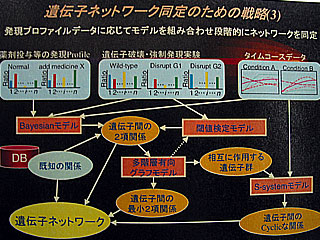
さて、遺伝子発現ネットワークの研究は、まずマイクロアレイなどの遺伝子発現プロファイルデータを用いて遺伝子間の相互作用を推定することからスタートする。そして、さまざまなネットワークモデルを当てはめ、目標の発現パターンに合致するネットワークの候補を洗い出すという手順をとる。しかし、一定の発現パターンに対して同じような挙動を示すネットワーク構造が複数存在するため、真のネットワークを同定するのは容易ではない。実際には、得られたネットワーク候補群をもとに次なる実験指針を立て、再度発現解析などを実施、その実験データを用いてさらにネットワーク候補を絞り込んでいくというルーチンワークが必要になる。 VoyaGeneはこうした研究プロセス全体を総合支援するシステムとして開発された。
その最大の特徴は、遺伝子ネットワーク解析の中核部分であるネットワークモデルに4種類のモデルを標準装備していること。具体的には、統計的な“しきい値検定モデル”、ベイズの定理に基づく“ベイジアンネットワークモデル”、時系列データから推定する“Sシステムモデル”、遺伝子間の二項関係から矛盾のないネットワークを導く“多階層有向グラフモデル”−の4種類をサポートしている。各モデルにはそれぞれ長所と短所があるが、VoyaGeneはそれらを段階的に組み合わせて使用することができるので、精度の高いネットワーク候補群を提示することが可能になる。製品化に向けては、さらに新しいモデルの導入も計画しているという。
なお、システム自体はクライアント/サーバー型で、サーバー側にはオープンソースのフリーデータベースシステム「PostgreSQL」(UNIXまたはLinux)を採用している。クライアント側はJavaが利用できればウィンドウズでもかまわない。試用版のダウンロードは以下のURL(http://voyagene.hydra.mki.co.jp/)から。