 東大医科研は、日本におけるゲノムを中心にした医学・生物医学研究の中核拠点である「ヒトゲノム解析センター」を有し、日本人の全ゲノム配列解析を世界で初めて行うなど、この分野の先進的な研究を推進してきている。とくに、次世代シーケンサー(NGS)の発達により大量のヒトゲノム情報を高速に取得できるようになったことを背景に、今回、個別化医療の実現に向けた研究を加速することを目的として、日立製作所の協力のもと、スーパーコンピューターシステムを刷新することになった。
東大医科研は、日本におけるゲノムを中心にした医学・生物医学研究の中核拠点である「ヒトゲノム解析センター」を有し、日本人の全ゲノム配列解析を世界で初めて行うなど、この分野の先進的な研究を推進してきている。とくに、次世代シーケンサー(NGS)の発達により大量のヒトゲノム情報を高速に取得できるようになったことを背景に、今回、個別化医療の実現に向けた研究を加速することを目的として、日立製作所の協力のもと、スーパーコンピューターシステムを刷新することになった。
東大医科研がヒトゲノム解析用スーパーコンピューターを刷新
日立製作所と共同で設置・稼働へ、トータル422TFLOPSの複合システム
2015.03.28−日立製作所は26日、東京大学医科学研究所において、ヒトゲノム解析用の新型スーパーコンピューター「Shirokane3」が4月1日から稼働すると発表した。理論最大性能は422テラFLOPSで、従来機に比べて約10倍の速度でヒトゲノムのデータ解析を行うことが可能。ストレージも拡張され、約100万人分のデータを保存することができる。これにより、ゲノムの変異した箇所を高速に検索・特定し、その変異と疾患との因果関係の分析や、治療効果の高い医薬品の予測を行うことなどが可能になる。
 東大医科研は、日本におけるゲノムを中心にした医学・生物医学研究の中核拠点である「ヒトゲノム解析センター」を有し、日本人の全ゲノム配列解析を世界で初めて行うなど、この分野の先進的な研究を推進してきている。とくに、次世代シーケンサー(NGS)の発達により大量のヒトゲノム情報を高速に取得できるようになったことを背景に、今回、個別化医療の実現に向けた研究を加速することを目的として、日立製作所の協力のもと、スーパーコンピューターシステムを刷新することになった。
東大医科研は、日本におけるゲノムを中心にした医学・生物医学研究の中核拠点である「ヒトゲノム解析センター」を有し、日本人の全ゲノム配列解析を世界で初めて行うなど、この分野の先進的な研究を推進してきている。とくに、次世代シーケンサー(NGS)の発達により大量のヒトゲノム情報を高速に取得できるようになったことを背景に、今回、個別化医療の実現に向けた研究を加速することを目的として、日立製作所の協力のもと、スーパーコンピューターシステムを刷新することになった。
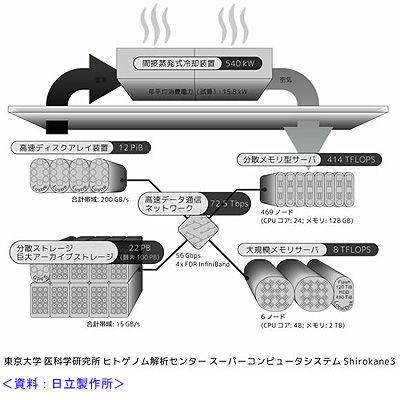
2012年1月から稼働している現行機の「Shirokane2」も併用され、新しい「Shirokane3」と合わせた全体の能力は、演算性能で600テラFLOPS、ストレージ容量で25ペタバイトに達する。
「Shirokane2」は日立製の「HA8000/RS210」(CPUはインテルのXeon)と「HA8000-tc/HT225」(CPUはAMDのOpteron)、SGIの「UV100」から構成される複合システムだが、今回の「Shirokane3」も同様に複合的な構成(別図参照)。8テラFLOPSの大規模共有メモリー型の計算サーバー(6ノード)は、デルの「PowerEdge R920」とHPの「ProLiant DL580 Gen8」から構成され、1ノード当たり2TBのメモリー、48個のCPUコア、1.6TBの内蔵フラッシュメモリーディスクを搭載している。一方の414テラFLOPSの分散メモリー型計算サーバーを構成するのは、HPの「HP Apollo 6000 SL230s Gen8 後継機」(445ノード)と「HP SL250s Gen8 後継機」(24ノード)で、このうちの後者はアクセラレーターとしてXeon Phi 7120Pを搭載したモデルになる。
ヒトゲノム情報の高速な並列処理を行うプログラム用には合計1万1,160コアを擁する分散メモリー型サーバーが、ヒトゲノムアセンブリープログラムといった大容量のメモリーを必要とする用途には1ノード当たり2TBのメモリーを持つ大規模メモリーサーバーが使用される。
また、膨大なヒトゲノム情報の高速な解析を行うため、すべてのサーバー間と大量のデータを高速に通信できる高速ディスクアレイ装置や、ヒトゲノム情報を格納するための大容量の分散ストレージと巨大アーカイブストレージが設置されており、全体で100ペタバイトを超える容量まで増設できるように設計されているという。
今回の「Shirokane3」は、環境に配慮した東京大学サステイナブルキャンパスプロジェクト(TSCP)の一環として省電力性も重視しており、国内の大学・研究機関では初となる間接蒸発式冷却装置を採用。室内の熱を外気と混ぜずに熱交換器を介して放出することにより、データセンターのエネルギー効率の指標であるPUE値(1に近いほどエネルギー効率が高い)で1.06という試算が得られている。
なお、今回の「Shirokane3」は、現行の「Shirokane2」と同様に国内の他の研究機関の研究者や医療関係者も利用することが可能。現在「Shirokane2」を利用中のグループは、4月末までの先行利用期間は特別な届出なしで「Shirokane3」を試すことができるという。
******
<関連リンク>:
東京大学医科学研究所ヒトゲノム解析センター(トップページ)
http://hgc.jp/japanese/
東京大学医科学研究所ヒトゲノム解析センター(スーパーコンピューターのページ)
https://supcom.hgc.jp/