 今回の研究は、抗ウイルス薬に限らず2,128種の既存医薬品の中から治療薬候補を探そうというもの。4種類の標的タンパク質に対して網羅的にドッキングシミュレーションを行っており、今回はその1つであるメインプロテアーゼ(Mpro)に対する結果を公表した。
今回の研究は、抗ウイルス薬に限らず2,128種の既存医薬品の中から治療薬候補を探そうというもの。4種類の標的タンパク質に対して網羅的にドッキングシミュレーションを行っており、今回はその1つであるメインプロテアーゼ(Mpro)に対する結果を公表した。
理研が「富岳」による新型コロナウイルス治療薬候補同定で中間報告
既存約2,000種をドッキングシミュレーション、高濃度MDで結合過程評価
2020.07.06−理化学研究所は3日、スーパーコンピューター「富岳」の先行利用(正式な共用開始は2021年度から)で文部科学省と連携して進めている新型コロナウイルス対策研究の一部成果を公表した。これは、京都大学大学院医学研究科の奥野恭史教授らによる「『富岳』による新型コロナウイルスの治療医薬候補同定」プロジェクトで、分子動力学(MD)シミュレーションを利用して既存医薬品データベースから新型コロナウイルスの標的タンパク質に高い親和性を示す治療薬候補を探索した。その結果、計算だけで数十種類の有望な候補を発見できたという。
 今回の研究は、抗ウイルス薬に限らず2,128種の既存医薬品の中から治療薬候補を探そうというもの。4種類の標的タンパク質に対して網羅的にドッキングシミュレーションを行っており、今回はその1つであるメインプロテアーゼ(Mpro)に対する結果を公表した。
今回の研究は、抗ウイルス薬に限らず2,128種の既存医薬品の中から治療薬候補を探そうというもの。4種類の標的タンパク質に対して網羅的にドッキングシミュレーションを行っており、今回はその1つであるメインプロテアーゼ(Mpro)に対する結果を公表した。
これまでのドッキングシミュレーションは、標的タンパク質の活性部位に薬物分子を配置し、その結合の強さを評価していた。今回は、薬物分子を離れた位置に置き、どのように結合するかをタンパク質を動かしながら計算する。このような手法は、スーパーコンピューター「京」でも十数個の化合物を評価するのが精一杯だったが、数千個の化合物を扱うことができれば世界初の成果になるという。
薬物の結合過程を十分に観察するには、最低でも1ミリ秒のMDシミュレーションが望ましいが、数千種類の化合物をこの時間スケールで計算することは「富岳」でも難しい。そこで、奥野教授らのグループは、標的タンパク質の周りに配置する薬物分子の濃度を数百倍にし、薬物とタンパク質の衝突頻度を上げることにより、短い時間スケールで結合過程をとらえられるようにした。MDプログラムは、こういった計算手法に対応しているGROMACSを採用した。
今回のMproに対する2,128種の既存薬のドッキングシミュレーション結果は別図の通り。縦軸は活性ポケットに対する結合の強さ、横軸はタンパク質の表面全体に対する結合の強さで、右上のエリアが有望と思われる物質が集まっている領域になる。この中で、すでに新型コロナに関する臨床研究、治験が実施されているものが12種類みつかった。このうち、寄生虫駆虫薬のニクロサミド(日本では未承認、米国でも一般販売されていない)は、実験でMpro阻害活性を有することが報告されている。また、C型肝炎治療薬のニタゾキサニドは新型コロナにも有効だという研究があるが、標的タンパク質は明らかになっていなかった。今回の結果から、これもMpro標的であることが示唆されたことになる。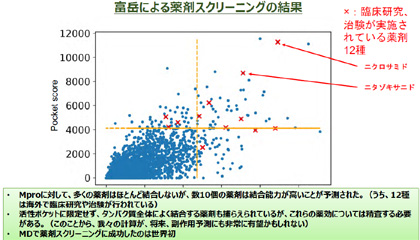
そのほかにも、まだ新型コロナへの適用が検討されていない既存薬が多数みつかっており、その中には国内の製薬会社がライセンスを持つ薬物も含まれるという。今後、それぞれのグループと臨床研究や治験について協議するほか、理研において細胞実験での検証を行うことも考えていく。さらに、残り3つの標的タンパク質に対するドッキングシミュレーションを進めることで新しい薬物が発見されれば、それらを併用することで治療効果が高まる可能性もあるということだ。
一方、横軸で評価したように、結合ポケット以外の場所に薬物が結合することは予期しない副作用につながることも考えられる。今回の計算手法は、薬効を評価するだけでなく、副作用の解明にも光を当てる可能性があるとしている。
今回、Mproに対するドッキングシミュレーションの実行に当たり、「富岳」で10日間を要した。ただ、使用したGROMACSは「富岳」に最適化されていない。「京」でも使用された国産MDソフトGENESISの「富岳」へのチューニングが進められており、GENESISに高濃度計算機能を搭載すれば、今回の計算は2日で完了したという。いずれにしても、数千個レベルの化合物とタンパク質の作用過程をMD計算した事例は世界でも初めてであり、学術的にも高いインパクトがあるとしている。
******
<関連リンク>:
理化学研究所(計算科学研究センターのトップページ)
https://www.r-ccs.riken.jp/jp/