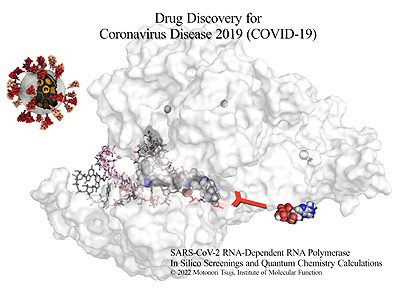
 今回の研究は、欧州バイオインフォマティクス研究所が管理運営する生物活性化合物データベース「ChEMBL」に登録されている約200万化合物を対象にした。新型コロナウイルスのRNA依存性RNAポリメラーゼ(RdRp)の活性中心に特異的に結合する物質を、ドッキングプログラムの「AutoDock Vina」および「rDock」を利用してバーチャルスクリーニングしたもの。
今回の研究は、欧州バイオインフォマティクス研究所が管理運営する生物活性化合物データベース「ChEMBL」に登録されている約200万化合物を対象にした。新型コロナウイルスのRNA依存性RNAポリメラーゼ(RdRp)の活性中心に特異的に結合する物質を、ドッキングプログラムの「AutoDock Vina」および「rDock」を利用してバーチャルスクリーニングしたもの。
分子機能研究所がコロナ感染症治療薬の候補物質をリスト化
全系量子化学計算で解析、有用性高い医薬品開発に道
2022.09.27−分子機能研究所はこのほど、最先端のバーチャルスクリーニングにより、新型コロナウイルス感染症治療薬の候補物質として、既存医薬品249品目と生物活性化合物184品目を見いだしたと発表した。辻一徳(つじもとのり)代表の研究成果で、International Journal of Molecular Sciences誌に論文が掲載された。ここ1年ほどは、実験での考察を含まないシミュレーションだけの論文は受理されないことがほとんどだというが、今回の研究は全系の量子化学計算を利用した手法の新規性と、予測結果の精度が実際に即していたことなどが評価され、掲載に至ったもよう。同社では、今回の成果をベースに広く共同研究先を募り、実験での評価を進めたいとしている。
 今回の研究は、欧州バイオインフォマティクス研究所が管理運営する生物活性化合物データベース「ChEMBL」に登録されている約200万化合物を対象にした。新型コロナウイルスのRNA依存性RNAポリメラーゼ(RdRp)の活性中心に特異的に結合する物質を、ドッキングプログラムの「AutoDock Vina」および「rDock」を利用してバーチャルスクリーニングしたもの。
今回の研究は、欧州バイオインフォマティクス研究所が管理運営する生物活性化合物データベース「ChEMBL」に登録されている約200万化合物を対象にした。新型コロナウイルスのRNA依存性RNAポリメラーゼ(RdRp)の活性中心に特異的に結合する物質を、ドッキングプログラムの「AutoDock Vina」および「rDock」を利用してバーチャルスクリーニングしたもの。
実際に標的としたのはRdRpに二重鎖RNAが結合した巨大生体高分子複合体で、クライオ電子顕微鏡データをもとに、同社が独自開発した「Homology Modeling for HyperChem」でモデリングした。サブユニット、活性中心に存在するマグネシウムイオン、複製中のRNA構造も含めてRdRp複合体すべてをモデル化し、トータル2万原子をGaussianのONIOM法で構造最適化した。独自のONIOMインターフェースを備えているため、量子力学(QM)で計算するハイレイヤーと分子力学(MM)で計算するローレイヤーの設定が容易であり、2万原子のQM/MM計算でも実行が可能だったという。また、Gaussianの振動解析によって結合自由エネルギーを算出。治療薬候補物質が標的(RdRp複合体)とどれくらい強く結合できるかの指標となる数値を予測した。
さらに、フラグメント分子軌道法(FMO)を利用し、RdRp複合体(1,163残基)に対して全系量子化学計算での一点計算(MP2法)を実施。候補化合物がRdRp複合体のどの残基と強く相互作用(静電相互作用、電子相関など)するかも解析した。このレベルのFMO計算に成功した例はこれまでにほとんどないという。どのように医薬化合物をデザインすれば、より強く相互作用するかのヒントになるため、有効な医薬品開発への示唆を与えるとして注目される。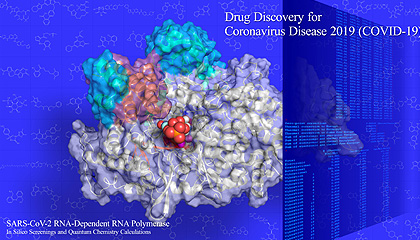
この結果、選び出された既存医薬品249品目には、レムデシビルやファビピラビル、ペンシクロビルなどの抗ウイルス薬も実際の実験結果を再現するかたちで含まれており、とくにペンシクロビルはこの論文が投稿されたあとに実際に結合することが報告されており、今回の予測結果の正しさを証明する例ともなっている。
一方、既存薬ではない生物活性化合物184品目は構造的特徴が類似しており、ほとんどがメジャーグルーブバインダーと呼ばれるウイルスRNAの溝にはまり込む仕組みを持っていた。これまでに承認されているコロナ治療薬は、標的と共有結合して不可逆的に結びつくことでウイルスの複製・増殖を阻害するメカニズムを持つが、これだといわゆる副作用が強い可能性がある。これに対し、メジャーグルーブバインダーは標的を可逆的に阻害する仕組みであり、副作用の原因にもなりにくい。有望な候補物質が含まれている可能性もありそうだ。
******
<関連リンク>:
分子機能研究所(トップページ)
https://www.molfunction.com/jp/
International Journal of Molecular Sciences誌(今回の論文へのリンク)
https://www.mdpi.com/1422-0067/23/19/11009/htm